工具集
突变工具
序列工具

ASO Designer
ASO 设计
通过计算与目标 RNA 的结合亲和力,对反义核苷酸进行设计
Sequence Alignment
序列比对
用于对比碱基或氨基酸序列相似性的工具
Reverse Complement Converter
反向互补转化
将输入序列转化为其反向,互补或者反向互补序列
Sequence Viewer
序列查看
查看特定基因的 cDNA, RNA 转录本和相应蛋白质序列
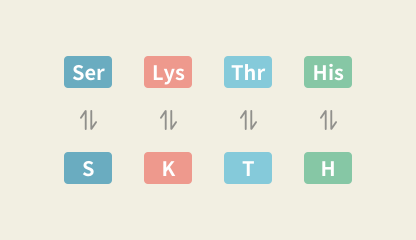
Amino Acid Symbol Converter
氨基酸简称转换工具
将氨基酸与其简称互相转换
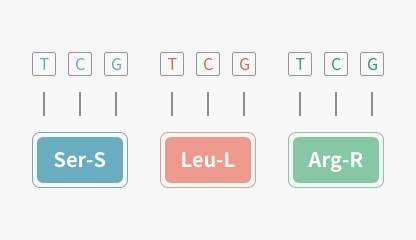
Sequence Translator
序列翻译
将 DNA 或 RNA 序列翻译为氨基酸序列
BLAT
BLAT
类BLAST工具:可快速找到相似度大于95%碱基序列
比对工具
其他工具
AI 工具

AI Assistant
AI助手
生物领域的专业AI助手
AI 工具
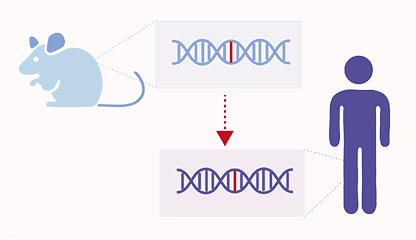
Homolog Predictor
同源基因预测
通过小鼠基因表达预测人类同源基因变化的 AI 工具
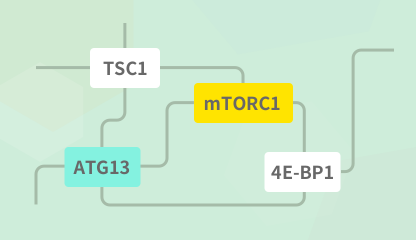
Pathway Enrichment Analysis
通路富集分析
对基因表达数据进行通路富集分析
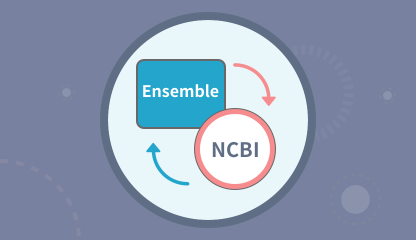
Ensembl-NCBI Converter
Ensembl-NCBI 转换
对 Ensembl 与 NCBI 的基因 ID 互相转换
AI 工具
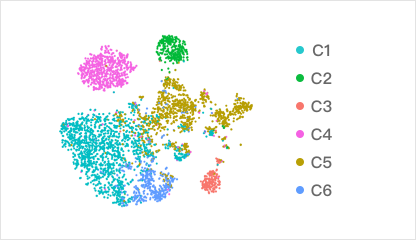
Gene Expression Database
基因表达可视化数据库
可查询特定基因表达的基于单细胞测序文献的可视化 AI 数据库
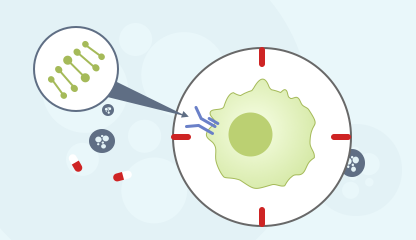
Cancer Target Database
癌症靶点数据库
查询特定癌症靶点对药物的反应
AI 工具
Affinity Predictor
亲和力预测
预测蛋白复合体结合亲和力的 AI 工具